
Pomiar różnorodności
Miary różnorodności
Papierkiem lakmusowym naporu selekcyjnego jest różnorodność populacji, dlatego powstała potrzeba jej mierzenia. Miary różnorodności zwykle dzieli się na genotypowe i fenotypowe. Te pierwsze informują nas o różnicach w zakodowanych rozwiązaniach, natomiast różnice fenotypowe określają różnice w wartości funkcji przystosowania.
Wadami miar genotypowych są: ich większa złożoność obliczeniowa oraz konieczność dostosowania miary do użytego kodowania. Koszt obliczeniowy miar fenotypowych jest niewielki, jednak są mniej dokładne - rozwiązania o podobnej wartości przystosowania należące do różnych minimów zostaną przez nie uznane za podobne.
Miary fenotypowe
Wśród miar fenotypowych można wymienić odchylenie standardowe wartości przystosowania [47], a także liczbę różnych wartości przystosowania [48].
Rosca w [32] proponuje użycie entropii wartości przystosowania w populacji. Niech pk oznacza stosunek liczby osobników posiadających wartość przystosowania k do liczby wszystkich osobników. Wtedy entropia układu wynosi:
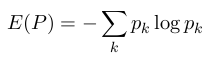
McPhee w pracy na temat Programowania Genetycznego [29] proponuje analizę historii populacji do badania stopnia wykorzystania początkowego bogactwa materiału genetycznego w końcowej populacji. Podobne rozwiązanie można spotkać w [47], gdzie podczas inicjalizacji każdemu osobnikowi nadaje się unikalny identyfikator Uid. W procesie krzyżowania jeden z potomków otrzymuje identyfikator pierwszego rodzica, a drugi potomek identyfikator drugiego rodzica.
Miary genotypowe
Najczęściej spotykaną miarą genotypową jest odległość Hamminga między dwoma rozwiązaniami. Z punktu widzenia niniejszej pracy miara ta posiada dwie zasadnicze wady. Po pierwsze miara ta porównuje wartości genów na absolutnych pozycjach (locus) a nie względnych. Można zastosować odległość Levenshteina, która bierze pod uwagę przestawienia i może być stosowana do problemu TSP, jednak obie metody wymagają wykonania (N - 1)! porównań, więc są zbyt obciążające obliczeniowo.
Inspiracją dla dalszych badań okazały się prace Morrisona i DeJonga [31] oraz Tsujimury i Gena [39], którzy zaproponowali dwa różne algorytmy o liniowej złożoności obliczeniowej O(N). Morrison i DeJong stworzyli nową miarę w oparciu o wzór na inercję n-wymiarowej bryły. Miara zdefinowana została w następujący sposób:
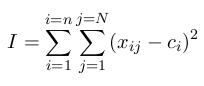
przy czym ci to współrzędna środka masy (centroidu), zaś xij oznacza wartość i-tego genu w j-tym chromosomie.
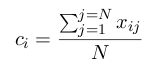
Autorzy dowodzą, że przy odpowiednim kodowaniu powyższa miara jest tożsama z miarą Hamminga, lecz od niej mniej złożona obliczeniowo.
Tsujimura i Gen, podobnie jak Rosca, proponują skorzystanie z praw entropii informacji dla rozwiązywania zagadnienia TSP. Jednak obie metody porównują wartości genów na absolutnych pozycjach. W rozdziale 3 zaproponowano nowy sposób liczenia różnorodności, który jest specyficzny dla zagadnienia permutacyjnego.
Obszerną analizę zagadnienia różnorodności oraz miar można także znaleźć w pracy doktorskiej S.M. Gustafsonao tytule An Analysis of Diversity in Genetic Programming ([16]).
Zastosowania
Mierzenie różnorodności populacji może mieć wiele zastosowań. Pierwszym z nich jest analiza wpływu poszczególnych parametrów na różnorodność. Zhu [47] bada wpływ następujących prawdopodobieństw: krzyżowania, mutacji oraz dostarczenia nowowygenerowanych osobników na różne miary różnorodności.
Drugim obszarem zastosowań jest użycie miary różnorodności jako czynnika wpływającego na przebieg algorytmu. Zhu w [47] proponuje następujący wzór na kontrolę współczynników mutacji i krzyżowania:
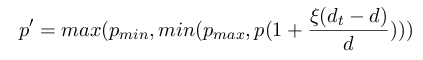
gdzie p to aktualna wartość współczynnika,P' - wartość współczynnika w kolejnej generacji, d -aktualna miara różnorodności, dt - docelowa miara różnorodności, a ξ - wrażliwość zmian. Dla unormowanej, genotypowej miary autor przyjmuje dt = 0,5.
Fukunaga z kolei proponuje strategię restartu, czyli ponownego rozpoczęcia ewolucji z nową populacją, w oparciu o historię poprzednich wykonań algorytmu [11].
komentarze
Copyright © 2008-2010 EPrace oraz autorzy prac.